7 Lihtne töö Andmeraamidega
7.1 Võrdleme andmeraame kahel viisil ja summeerime andmeraami.
- all_equal
df1 on märklaud ja df2 on see, mida võrreldakse. convert = TRUE ühtlustab kahe tabeli vahel sarnased andmetüübid (n. factor ja character).
all_equal(df1, df2, convert = FALSE)- diffdf raamatukogu annab detailsema väljundi
diffdf::diffdf(df1, df2)Andmetabeli Summary saab mitmel viisil, skimr::skim() funktsioon on üks paremaid
skimr::skim(iris)
#> Skim summary statistics
#> n obs: 150
#> n variables: 5
#>
#> ── Variable type:factor ────────────────────────────────────────────────────────
#> variable missing complete n n_unique
#> Species 0 150 150 3
#> top_counts ordered
#> set: 50, ver: 50, vir: 50, NA: 0 FALSE
#>
#> ── Variable type:numeric ───────────────────────────────────────────────────────
#> variable missing complete n mean sd p0 p25
#> Petal.Length 0 150 150 3.76 1.77 1 1.6
#> Petal.Width 0 150 150 1.2 0.76 0.1 0.3
#> Sepal.Length 0 150 150 5.84 0.83 4.3 5.1
#> Sepal.Width 0 150 150 3.06 0.44 2 2.8
#> p50 p75 p100
#> 4.35 5.1 6.9
#> 1.3 1.8 2.5
#> 5.8 6.4 7.9
#> 3 3.3 4.4BaasR kasutab summary(df) vormi.
7.2 Põhitehted andmeraamidega
count(iris, Species) #loeb üles, mitu korda igat näitu veerus Species esineb
#> # A tibble: 3 x 2
#> Species n
#> <fct> <int>
#> 1 setosa 50
#> 2 versicolor 50
#> 3 virginica 50
summary(iris)
#> Sepal.Length Sepal.Width Petal.Length
#> Min. :4.30 Min. :2.00 Min. :1.00
#> 1st Qu.:5.10 1st Qu.:2.80 1st Qu.:1.60
#> Median :5.80 Median :3.00 Median :4.35
#> Mean :5.84 Mean :3.06 Mean :3.76
#> 3rd Qu.:6.40 3rd Qu.:3.30 3rd Qu.:5.10
#> Max. :7.90 Max. :4.40 Max. :6.90
#> Petal.Width Species
#> Min. :0.1 setosa :50
#> 1st Qu.:0.3 versicolor:50
#> Median :1.3 virginica :50
#> Mean :1.2
#> 3rd Qu.:1.8
#> Max. :2.5
names(iris) #annab veerunimed
#> [1] "Sepal.Length" "Sepal.Width" "Petal.Length"
#> [4] "Petal.Width" "Species"
nrow(iris) #mitu rida?
#> [1] 150
ncol(iris) #mitu veergu?
#> [1] 5
arrange(iris, desc(Sepal.Length)) %>% head(3)
#> Sepal.Length Sepal.Width Petal.Length Petal.Width
#> 1 7.9 3.8 6.4 2.0
#> 2 7.7 3.8 6.7 2.2
#> 3 7.7 2.6 6.9 2.3
#> Species
#> 1 virginica
#> 2 virginica
#> 3 virginica
#sorteerib tabeli veeru "Sepal.Length" väärtuste järgi
#langevalt (default on tõusev sorteerimine).
#Võib argumendina anda mitu veergu.
top_n(iris, 2, Sepal.Length)
#> Sepal.Length Sepal.Width Petal.Length Petal.Width
#> 1 7.7 3.8 6.7 2.2
#> 2 7.7 2.6 6.9 2.3
#> 3 7.7 2.8 6.7 2.0
#> 4 7.9 3.8 6.4 2.0
#> 5 7.7 3.0 6.1 2.3
#> Species
#> 1 virginica
#> 2 virginica
#> 3 virginica
#> 4 virginica
#> 5 virginica
#saab 2 või rohkem rida, milles on kõige suuremad S.L. väärtused
top_n(iris, -2, Sepal.Length) #saab 2 rida, milles on kõige väiksemad väärtused
#> Sepal.Length Sepal.Width Petal.Length Petal.Width
#> 1 4.4 2.9 1.4 0.2
#> 2 4.3 3.0 1.1 0.1
#> 3 4.4 3.0 1.3 0.2
#> 4 4.4 3.2 1.3 0.2
#> Species
#> 1 setosa
#> 2 setosa
#> 3 setosa
#> 4 setosaTibblega saab teha maatriksarvutusi, kui kasutada ainult arvudega ridu. apply() arvutab maatriksi rea (1) või veeru (2) kaupa, vastavalt funktsioonile, mille sa ette annad.
colSums(fruits[ , 2:3])
#> apples oranges
#> 48 NA
rowSums(fruits[ , 2:3])
#> [1] 3 36 NA
rowMeans(fruits[ , 2:3])
#> [1] 1.5 18.0 NA
colMeans(fruits[ , 2:3])
#> apples oranges
#> 16 NA
fruits_subset <- fruits[ , 2:3]
# 1 tähendab, et arvuta sd rea kaupa
apply(fruits_subset, 1, sd)
#> [1] 0.707 19.799 NA
# 2 tähendab, et arvuta sd veeru kaupa
apply(fruits_subset, 2, sd)
#> apples oranges
#> 23.4 NALisame käsitsi tabelile rea:
fruits <- add_row(fruits,
shop = "konsum",
apples = 132,
oranges = -5,
.before = 3)
fruits
#> # A tibble: 4 x 4
#> shop apples oranges vabakava
#> <chr> <dbl> <dbl> <list>
#> 1 maxima 1 2 <chr [26]>
#> 2 tesco 4 32 <dbl [10]>
#> 3 konsum 132 -5 <NULL>
#> 4 lidl 43 NA <lm>Proovi ise:
add_column()Eelnevaid verbe ei kasuta me just sageli, sest tavaliselt loeme andmed sisse väljaspoolt R-i. Aga väga kasulikud on järgmised käsud:
7.3 Rekodeerime andmeraami väärtusi
fruits$apples[fruits$apples==43] <- 333
fruits
#> # A tibble: 4 x 4
#> shop apples oranges vabakava
#> <chr> <dbl> <dbl> <list>
#> 1 maxima 1 2 <chr [26]>
#> 2 tesco 4 32 <dbl [10]>
#> 3 konsum 132 -5 <NULL>
#> 4 lidl 333 NA <lm>
fruits$shop[fruits$shop=="tesco"] <- "TESCO"
fruits
#> # A tibble: 4 x 4
#> shop apples oranges vabakava
#> <chr> <dbl> <dbl> <list>
#> 1 maxima 1 2 <chr [26]>
#> 2 TESCO 4 32 <dbl [10]>
#> 3 konsum 132 -5 <NULL>
#> 4 lidl 333 NA <lm>
fruits$apples[fruits$apples>100] <- NA
fruits
#> # A tibble: 4 x 4
#> shop apples oranges vabakava
#> <chr> <dbl> <dbl> <list>
#> 1 maxima 1 2 <chr [26]>
#> 2 TESCO 4 32 <dbl [10]>
#> 3 konsum NA -5 <NULL>
#> 4 lidl NA NA <lm>Viskame välja duplikaatread, aga ainult need kus veerg nimega col1 sisaldab identseid väärtusi (mitmest identse väärtusega reast jääb alles ainult esimene)
distinct(dat, col1, .keep_all = TRUE)
# kõikide col vastu
distinct(dat) Rekodeerime Inf ja NA väärtused nulliks (mis küll tavaliselt on halb mõte):
# inf to 0
x[is.infinite(x)] <- 0
# NA to 0
x[is.na(x)] <- 07.4 Ühendame kaks andmeraami rea kaupa
Tabeli veergude arv ei muutu, ridade arv kasvab.
dfs <- tibble(colA = c("a", "b", "c"), colB = c(1, 2, 3))
dfs1 <- tibble(colA = "d", colB = 4)
#id teeb veel ühe veeru, mis näitab, kummast algtabelist iga uue tabeli rida pärit on
bind_rows(dfs, dfs1, .id = "id")
#> # A tibble: 4 x 3
#> id colA colB
#> <chr> <chr> <dbl>
#> 1 1 a 1
#> 2 1 b 2
#> 3 1 c 3
#> 4 2 d 4Vaata Environmentist need tabelid üle ja mõtle järgi, mis juhtus.
Kui bind_rows() miskipärast ei tööta, proovi do.call(rbind, dfs), mis on väga sarnane.
NB! Alati kontrollige, et ühendatud tabel oleks selline, nagu te tahtsite!
Näiteks, võib-olla te tahtsite järgnevat tabelit saada, aga võib-olla ka mitte:
df2 <- tibble(ColC = "d", ColD = 4)
## works by guessing your true intention
bind_rows(dfs1, df2)
#> # A tibble: 2 x 4
#> colA colB ColC ColD
#> <chr> <dbl> <chr> <dbl>
#> 1 d 4 <NA> NA
#> 2 <NA> NA d 47.5 ühendame kaks andmeraami veeru kaupa
Meil on 2 verbi: bind_cols ja cbind, millest esimene on konservatiivsem. Proovige eelkõige bind_col-ga läbi saada, aga kui muidu ei saa, siis cbind ühendab vahest asju, mida bind_cols keeldub puutumast. NB! Alati kontrollige, et ühendatud tabel oleks selline, nagu te tahtsite!
dfx <- tibble(colC = c(4, 5, 6))
bind_cols(dfs, dfx)
#> # A tibble: 3 x 3
#> colA colB colC
#> <chr> <dbl> <dbl>
#> 1 a 1 4
#> 2 b 2 5
#> 3 c 3 67.6 andmeraamide ühendamine join()-ga
Kõigepealt 2 tabelit: df1 ja df2.
df1 <- tribble(
~ Member, ~ yr_of_birth,
"John Lennon", 1940,
"Paul McCartney", 1942
)
df1
#> # A tibble: 2 x 2
#> Member yr_of_birth
#> <chr> <dbl>
#> 1 John Lennon 1940
#> 2 Paul McCartney 1942df2 <- tribble(
~ Member, ~ instrument, ~ yr_of_birth,
"John Lennon", "guitar", 1940,
"Ringo Starr", "drums", 1940,
"George Harrisson", "guitar", 1942
)
df2
#> # A tibble: 3 x 3
#> Member instrument yr_of_birth
#> <chr> <chr> <dbl>
#> 1 John Lennon guitar 1940
#> 2 Ringo Starr drums 1940
#> 3 George Harrisson guitar 1942Ühendan 2 tabelit nii, et mõlema tabeli kõik read ilmuvad uude tabelisse.
full_join(df1, df2)
#> # A tibble: 4 x 3
#> Member yr_of_birth instrument
#> <chr> <dbl> <chr>
#> 1 John Lennon 1940 guitar
#> 2 Paul McCartney 1942 <NA>
#> 3 Ringo Starr 1940 drums
#> 4 George Harrisson 1942 guitarÜhendan esimese tabeliga df2 nii, et ainult df1 read säilivad, aga df2-lt võetakse sisse veerud, mis df1-s puuduvad. See on hea join, kui on vaja algtabelile lisada infot teistest tabelitest.
left_join(df1, df2)
#> # A tibble: 2 x 3
#> Member yr_of_birth instrument
#> <chr> <dbl> <chr>
#> 1 John Lennon 1940 guitar
#> 2 Paul McCartney 1942 <NA>Jätan alles ainult need df1 read, millele vastab mõni df2 rida.
semi_join(df1, df2)
#> # A tibble: 1 x 2
#> Member yr_of_birth
#> <chr> <dbl>
#> 1 John Lennon 1940Jätan alles ainult need df1 read, millele ei vasta ükski df2 rida.
anti_join(df1, df2)
#> # A tibble: 1 x 2
#> Member yr_of_birth
#> <chr> <dbl>
#> 1 Paul McCartney 19427.7 Nii saab raamist kätte vektori, millega tehteid teha.
Tibble jääb muidugi endisel kujul alles.
ubinad <- fruits$apples
ubinad <- ubinad + 2
ubinad
#> [1] 3 6 NA NA
## see on jälle vektor
str(ubinad)
#> num [1:4] 3 6 NA NA7.8 Andmeraamide salvestamine (eksport-import)
Andmeraami saame salvestada näiteks csv-na (comma separated file) oma kõvakettale, kasutame “tidyverse” analooge paketist “readr”, mille nimed on baas R funktsioonidest eristatavad alakriipsu “_" kasutamisega. “readr” laaditakse automaatselt koos “tidyverse” laadimisega.
## loome uuesti fruits data tibble
shop <- c("maxima", "tesco", "lidl")
apples <- c(1, 4, 43)
oranges <- c(2, 32, NA)
fruits <- tibble(shop, apples, oranges, vabakava)
## kirjutame fruits tabeli csv faili fruits.csv kataloogi data
write_csv(fruits, "data/fruits.csv")Kuhu see fail läks? See läks meie projekti juurkataloogi kausta “data/”, juurkataloogi asukoha oma arvuti kõvakettal leiame käsuga:
getwd()
#> [1] "/Users/ulomaivali/Dropbox/loengud/R course/2017- R course/loengu-rmd-konspektid"Andmete sisselugemine töökataloogist:
fruits <- read_csv("data/fruits.csv")Andmeraamide sisselugemiseks on kaks paralleelset süsteemi: baas-R-i read.table() ja selle mugavusfunktsioonid (read.csv(), read.csv2() jne) ning readr paketti, mis laaditakse koos tidyversiga, funktsioon read_delim() ja selle mugavusfunktsioonid (read_csv jne). Tavaliselt soovitame eelistada alakriipsuga variante (http://r4ds.had.co.nz/data-import.html).
read_delim()-l ja selle poegade argument col_types = cols(col_name_1 = col_double(), col_name_2 = col_date(format = "")) võimaldab spetsifitseerida kindlatele veergudele, mis tüübiga need sisse loetakse. Töötab ka cols_only(a = col_integer()), samuti standardsed lühendid andmetüüpidele: cols(a = "i", b = "d"). Vaikimisi otsustab programm andmetüübi iga veeru esimese 1000 elemendi põhjal. Vahest tasub kõik veerud sisse lugeda character-idena, et oleks parem probleeme tuvastada: df1 <- read_csv("my_data_frame_name.csv"), col_types = cols(.default = col_character())). .default - kõik nimega veerud, mille kohta ei ole eksplitsiitselt teisiti õeldud, lähevad sisse lugemisel selle alla. .
Seda, milline sümbol kodeerib sisseloetavas failis koma ja milline on “grouping mark”, mis eraldab tuhandeid, saab sisestada locale = locale(decimal_mark = ",", grouping_mark = ".") abil. Või näit: locale("et", decimal_mark = ";"). Vt ka https://cran.r-project.org/web/packages/readr/vignettes/locales.html.
https://en.wikipedia.org/wiki/List_of_ISO_639-1_codes annab nimekirja riikide lokaalitähistest.
Argument skip = n jätab esimesed n rida sisse lugemata. Argument comment = "#" jätab sisse lugemata read, mis algavad #-ga.
Argument col_names = FALSE ei loe esimest rida sisse veerunimedena (see on vaikekäitumine) ja selle asemel nimetatakse veerud X1 … Xn. col_names = c("x", "y", "z")) loeb tabeli sisse uute veerunimedega x, y ja z.
na = "." argument ütleb, et tabeli kirjed, mis on punktid, tuleb sisse lugeda NA-dena.
Kui teil on kataloogitäis faile (näit .csv lõpuga), mida soovite kõiki korraga sisse lugeda, siis tehke nii:
library(fs)
#järgnev loeb sisse iga faili eraldi kataloogist nimega data_dir
fs::dir_ls(data_dir, regexp = "\\.csv$") %>% map(read_csv)
#Kui meil on mitu faili samade tulbanimedega ja tahame
#need sisse lugeda ühte faili üksteise järel, siis
dir_ls(data_dir, regexp = "\\.csv$") %>% map_dfr(read_csv, .id = "source")
#.id on optsionaalne argument, mis lisab uude faili lisaveeru,
#kus on unikaalsed viited igale algtabelile, et oleks näha, millisest
#tabelist iga uue tabeli rida pärit on.MS exceli failist saab tabeleid importida “readxl” raamatukogu abil.
library(readxl)
## kõigepealt vaatame kui palju sheete failis on
sheets <- excel_sheets("data/excelfile.xlsx")
## siis impordime näiteks esimese sheeti
dfs <- read_excel("data/excelfile.xlsx", sheet = sheets[1])Excelist csv-na eksporditud failid tuleks sisse lugeda käsuga read_csv2 või read.csv2 (need on erinevad funktsioonid; read.csv2 loeb selle sisse data frame-na ja read_csv2 tibble-na).
R-i saab sisse lugeda palju erinevaid andmeformaate. Näiteks, installi RStudio addin: “Gotta read em all R”, vaata eespool. See läheb ülesse tab-i Addins. Sealt saab selle avada ja selle abil tabeleid oma workspace üles laadida.
Alternatiiv: mine alla paremake Files tab-le, navigeeri sinna kuhu vaja ja kliki faili nimele, mida tahad R-i importida.
Mõlemal juhul ilmub alla konsooli (all vasakul) koodijupp, mille jooksutamine peaks asja ära tegema. Te võite tahta selle koodi kopeerida üles vasakusse aknasse kus teie ülejäänud kood tulevastele põlvedele säilub.
Tüüpiliselt töötate R-s oma algse andmestikuga. Reprodutseeruvaks projektiks on vaja 2 asja: algandmeid ja koodi, millega neid manipuleerida. R ei muuda algandmeid, mille te näiteks csv-na sisse loete.
Andmetabelite salvestamine töö vaheproduktidena ei ole sageli vajalik, sest te jooksutate iga kord, kui te oma projekti juurde naasete, kogu analüüsi uuesti kuni kohani, kuhu te pooleli jäite. See tagab, et teie kood töötab tervikuna. Erandiks on tabelid, mille arvutamine võtab palju aega.
Tibble konverteerimine data frame-ks ja tagasi tibbleks:
class(fruits) #näitab ojekti klassi
#> [1] "tbl_df" "tbl" "data.frame"
fruits <- as.data.frame(fruits)
class(fruits)
#> [1] "data.frame"
fruits <- as_tibble(fruits)
class(fruits)
#> [1] "tbl_df" "tbl" "data.frame"7.9 NA-d
Loe üles NA-d, 0-d, inf-id ja unikaalsed väärtused.
library(funModeling)
diabetes <- read_delim("data/diabetes.csv",
";", escape_double = FALSE, trim_ws = TRUE)
diabetes %>% status() %>%
mutate_if(is.numeric, round, 2) %>%
kableExtra::kable()| variable | q_zeros | p_zeros | q_na | p_na | q_inf | p_inf | type | unique |
|---|---|---|---|---|---|---|---|---|
| id | 0 | 0 | 0 | 0.00 | 0 | 0 | numeric | 403 |
| chol | 0 | 0 | 1 | 0.00 | 0 | 0 | numeric | 154 |
| stab.glu | 0 | 0 | 0 | 0.00 | 0 | 0 | numeric | 116 |
| hdl | 0 | 0 | 1 | 0.00 | 0 | 0 | numeric | 77 |
| ratio | 0 | 0 | 1 | 0.00 | 0 | 0 | numeric | 69 |
| glyhb | 0 | 0 | 13 | 0.03 | 0 | 0 | numeric | 239 |
| location | 0 | 0 | 0 | 0.00 | 0 | 0 | character | 2 |
| age | 0 | 0 | 0 | 0.00 | 0 | 0 | numeric | 68 |
| gender | 0 | 0 | 0 | 0.00 | 0 | 0 | character | 2 |
| height | 0 | 0 | 5 | 0.01 | 0 | 0 | numeric | 22 |
| weight | 0 | 0 | 1 | 0.00 | 0 | 0 | numeric | 140 |
| frame | 0 | 0 | 12 | 0.03 | 0 | 0 | character | 3 |
| bp.1s | 0 | 0 | 5 | 0.01 | 0 | 0 | numeric | 71 |
| bp.1d | 0 | 0 | 5 | 0.01 | 0 | 0 | numeric | 57 |
| bp.2s | 0 | 0 | 262 | 0.65 | 0 | 0 | numeric | 48 |
| bp.2d | 0 | 0 | 262 | 0.65 | 0 | 0 | numeric | 36 |
| waist | 0 | 0 | 2 | 0.00 | 0 | 0 | numeric | 30 |
| hip | 0 | 0 | 2 | 0.00 | 0 | 0 | numeric | 32 |
| time.ppn | 0 | 0 | 3 | 0.01 | 0 | 0 | numeric | 60 |
diabetes <- read.table(file = "data/diabetes.csv", sep = ";", dec = ",", header = TRUE)
str(diabetes)
#> 'data.frame': 403 obs. of 19 variables:
#> $ id : int 1000 1001 1002 1003 1005 1008 1011 1015 1016 1022 ...
#> $ chol : int 203 165 228 78 249 248 195 227 177 263 ...
#> $ stab.glu: int 82 97 92 93 90 94 92 75 87 89 ...
#> $ hdl : int 56 24 37 12 28 69 41 44 49 40 ...
#> $ ratio : num 3.6 6.9 6.2 6.5 8.9 ...
#> $ glyhb : num 4.31 4.44 4.64 4.63 7.72 ...
#> $ location: Factor w/ 2 levels "Buckingham","Louisa": 1 1 1 1 1 1 1 1 1 1 ...
#> $ age : int 46 29 58 67 64 34 30 37 45 55 ...
#> $ gender : Factor w/ 2 levels "female","male": 1 1 1 2 2 2 2 2 2 1 ...
#> $ height : int 62 64 61 67 68 71 69 59 69 63 ...
#> $ weight : int 121 218 256 119 183 190 191 170 166 202 ...
#> $ frame : Factor w/ 4 levels "","large","medium",..: 3 2 2 2 3 2 3 3 2 4 ...
#> $ bp.1s : int 118 112 190 110 138 132 161 NA 160 108 ...
#> $ bp.1d : int 59 68 92 50 80 86 112 NA 80 72 ...
#> $ bp.2s : int NA NA 185 NA NA NA 161 NA 128 NA ...
#> $ bp.2d : int NA NA 92 NA NA NA 112 NA 86 NA ...
#> $ waist : int 29 46 49 33 44 36 46 34 34 45 ...
#> $ hip : int 38 48 57 38 41 42 49 39 40 50 ...
#> $ time.ppn: int 720 360 180 480 300 195 720 1020 300 240 ...
VIM::aggr(diabetes, prop = FALSE, numbers = TRUE)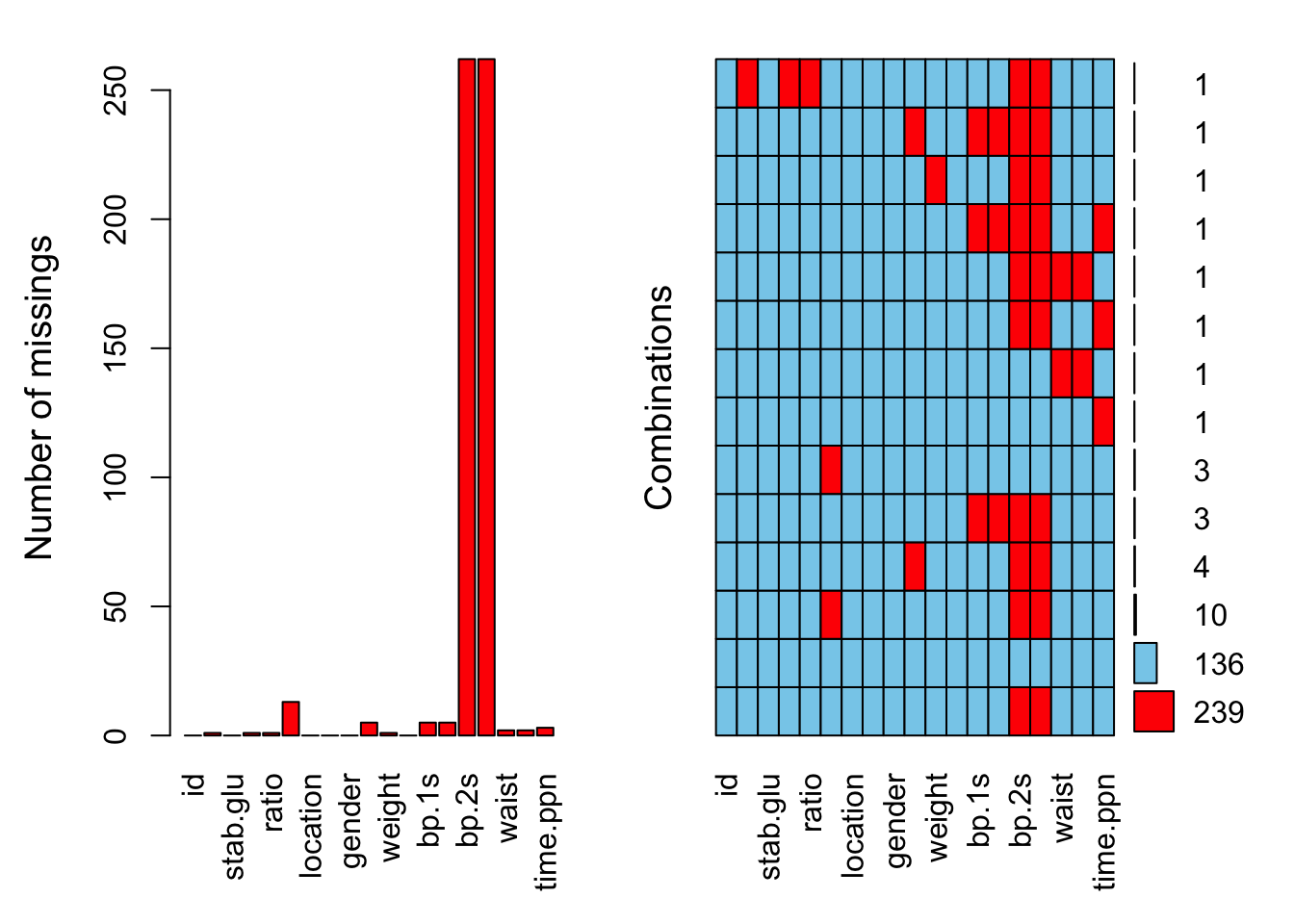 Siit on näha, et kui me viskame välja 2 tulpa ja seejärel kõik read, mis sisaldavad NA-sid, kaotame me umbes 20 rida 380-st, mis ei ole suur kaotus.
Siit on näha, et kui me viskame välja 2 tulpa ja seejärel kõik read, mis sisaldavad NA-sid, kaotame me umbes 20 rida 380-st, mis ei ole suur kaotus.
Kui palju ridu, milles on 0 NA-d? Mitu % kõikidest ridadest?
nrows <- nrow(diabetes)
ncomplete <- sum(complete.cases(diabetes))
ncomplete #136
#> [1] 136
ncomplete/nrows #34%
#> [1] 0.3377.9.1 Mitu NA-d on igas tulbas?
diabetes %>% map_df(~sum(is.na(.))) %>% t()
#> [,1]
#> id 0
#> chol 1
#> stab.glu 0
#> hdl 1
#> ratio 1
#> glyhb 13
#> location 0
#> age 0
#> gender 0
#> height 5
#> weight 1
#> frame 0
#> bp.1s 5
#> bp.1d 5
#> bp.2s 262
#> bp.2d 262
#> waist 2
#> hip 2
#> time.ppn 3väljund on uus tabel NA-de arvuga igale algse tabeli veerule
Eelnev ekspressioon töötab nii: map_df() loeb kokku (summeerib) diabetes tabeli igale veerule, mitu elementi selles veerus andis ekspressioonile is.na() vastuseks TRUE. is.na() on funktsioon, mis annab väljundiks TRUE v FALSE, sõltuvalt vektori elemendi NA-staatusest.
is.na(c(NA, "3", "sd", "NA"))
#> [1] TRUE FALSE FALSE FALSEPane tähele, et string “NA” ei ole sama asi, mis loogiline konstant NA.
Ploti NAd punasega igale tabeli reale ja tulbale mida tumedam halli toon seda suurem number selle tulba kontekstis:
VIM::matrixplot(diabetes)
#>
#> Click in a column to sort by the corresponding variable.
#> To regain use of the VIM GUI and the R console, click outside the plot region.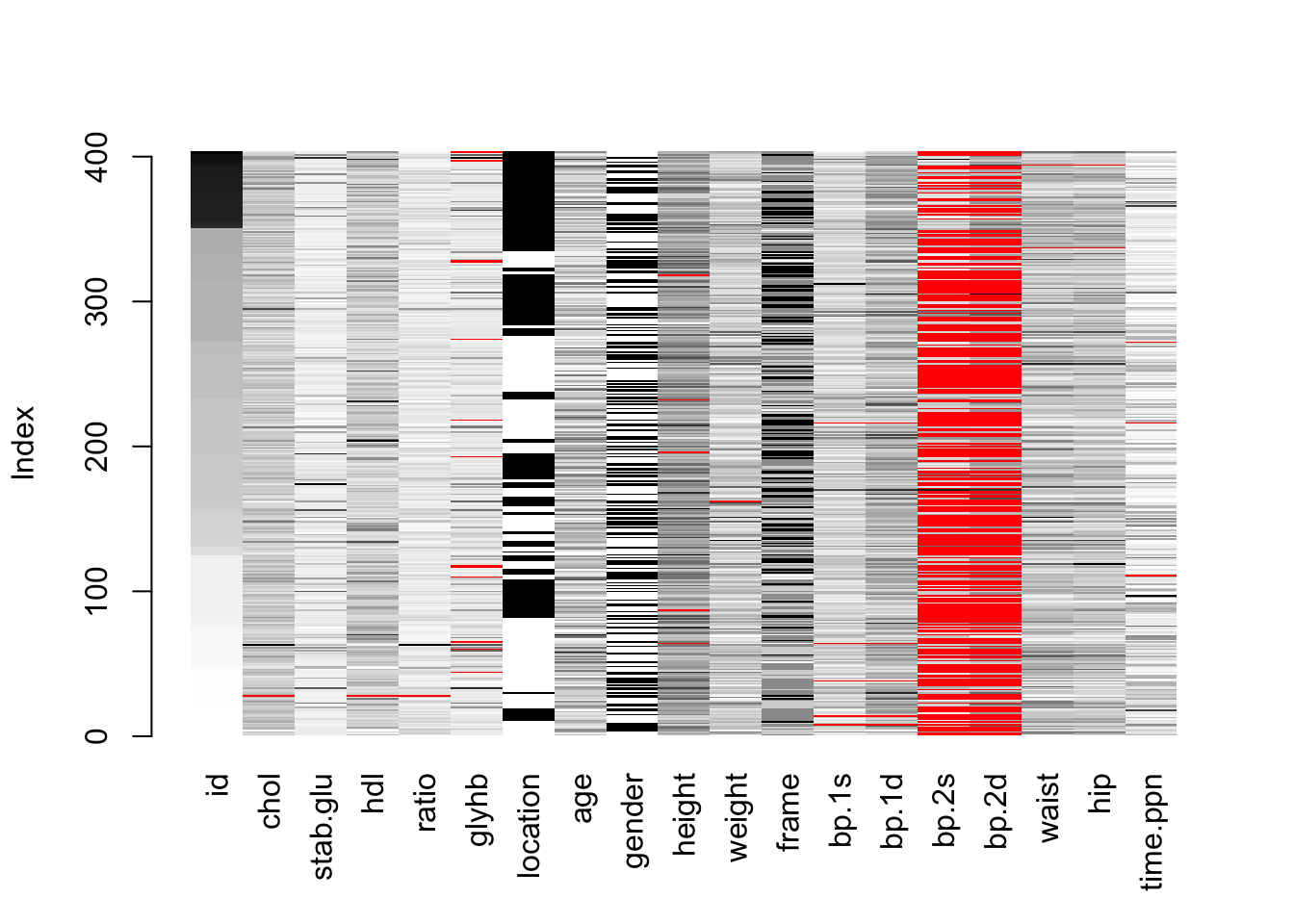
7.9.2 Kuidas rekodeerida NA-d näiteks 0-ks:
dfs[is.na(dfs)] <- 0
dfs[is.na(dfs)] <- "other"
dfs[dfs == 0] <- NA # teeb vastupidi 0-d NA-deksPane tähele, et NA tähistamine ei käi character vectorina vaid dedikeeritud is.na() funktsiooniga.
coalesce teeb seda peenemalt. kõigepealt kõik
x <- c(1:5, NA, NA, NA)
coalesce(x, 0L)
#> [1] 1 2 3 4 5 0 0 0Nii saab 2 vektori põhjal kolmanda nii, et NA-d asendatakse vastava väärtusega:
y <- c(1, 2, NA, NA, 5)
z <- c(NA, NA, 3, 4, 5)
coalesce(y, z)
#> [1] 1 2 3 4 5filter_all(weather, any_vars(is.na(.))) näitab ridu, mis sisaldavad NA-sid
filter_at(weather, vars(starts_with("wind")), all_vars(is.na(.))) read, kus veerg, mis sisaldab wind, on NA.
7.9.3 Rekodeerime NA-ks
na_if(x, y)
#x - vektor ehk tabeli veerg, mida modifitseerime
#y - väärtus, mida soovime NA-ga asendada
na_if(dfs, "") #teeb dfs tabelis tühjad lahtrid NA-deks
na_if(dfs, "other") #teeb lahtrid, kus on "other" NA-deks
na_if(dfs, 0) #teeb 0-d NA-deks. 7.9.4 drop_na() viskab tabelist välja NA-dega read
drop_na(data, c(column1, column2)) - argument variable võimaldab visata välja read, mis on NA-d ainult kindlates veergudes (column1 ja column2 meie näites - aga ära unusta column1 asemele kirjutada oma veeru nime). Ära unusta ka kasutamast vektori vormi c(), et veerge määrata. column1:column4 vorm töötab samuti ja võtab NAd veergudest 1-4 (kui veeru nr 1 nimi on column1 jne).
7.9.5 viska tabelist välja veerud, milles on liiga palju NA-sid
Meil on lai tabel sadade numbriliste veegudega. Neist paljud on NA-rikkad (andmevaesed) ja tuleks tabelist eemaldada. Aga kuidas seda teha?
Selleks (1) koostame vektori (vekt), milles on tabeli df-i iga numbrilise veeru NA-de suhtarv, (2) viskame sellest vektorist välja nende veergude nimed, milles on liiga palju NA-sid ja (3) subsetime df-i saadud vektori (vekt1) vastu.
NB! vekt ja vekt1 on nn named vektorid, milles vektori iga element (NAde suhtarv mingis tabeli veerus) on seotud selle elemendi nimega, mis on identne selle veeru nimega tabelis df.
vekt <- sapply(df, function(x) mean(is.na(x)))
#NA-de protsent igas veerus
vekt
vekt1 <- vekt[vekt < 0.8]
#subsettisin vektori elemendid, mis on < 0.8 (NA-sid alla 80%). 216 tk.
#vekt1 is a named vector
vekt1n <- names(vekt1) #vektor named vektori vekt1 nimedest
df_with_fewer_cols <- subset(df, select = vekt1n)
#subsetime (jätame alles) ainult need df-i veerud,
#mille nimele vastab mõni vektori nad1n element